果壳网已完,不知道我发过的帖子什么时候会消失,于是我要把它们搬到简书。
(注:在搬到新的博客的时候,这个帖子,以及整个果壳小组,都已经消失了。)
但这篇不是搬运,只是想说一下这个帖子背后的故事……以及代码。
Wolfram|Alpha 在显示计算或查询的结果之前,往往需要思考一番。几年前,它在思考过程的中会显示一个漂亮的图案。现在那个图案已经变成了一个无聊的 COMPUTING...,不过网上还是留下了一点痕迹,比如说知乎的这个问题:Wolfram|Alpha在搜索时出现的图案有什么含义?和 StackOverflow 的这个问题:What is the cellular automaton shown as loading screen on Wolfram Alpha?
我也一直想知道这是什么图案。它显然是一个元胞自动机,大小不同的点表示不同的状态;而且状态不止一种,因此肯定不是生命游戏,也不是生命游戏家族(Life-like)的规则。但它思考的速度太快,我总是来不及看清它到底是什么规则。
直到有一天,我在人人网上——当时人人网还不是直播平台——看到这么一个截图:

不知那位同学用的是什么截图工具,能够截得这么清晰,而且没有掉帧。总之,有了这个截图,我就能把它导入到 Mathematica,一帧一帧地慢慢看……并不能看出它是什么规则,甚至不能看清它有几种状态——最小的两种点个头差不多,只是颜色的深浅稍稍有点区别,肉眼不容易看清。不过我还是看出了一点规律:稍大一点的点会变得越来越大,直到爆掉,消失。而且,我还看到了这个一个振荡子:

除了 Life-like 的规则,还有什么规则?我在 Golly (最好的元胞自动机模拟器,没有之一)的帮助里看到了一类叫 Generations 的规则,觉得和这个有点像。不过那里给的几个 Generations 规则的例子和 Wolfram|Alpha 的这个元胞自动机都对不上。
Generations 的规则和 Life-like 的规则类似,每个细胞也有死活两种状态,下一回合的状态由这一回合的死活、以及与它相邻的活细胞的个数来决定。与 Life-like 不同的是,活细胞在临死之前,还能苟延残喘几个回合,但死亡的过程并不受周围的细胞影响,最终也无法摆脱死亡的命运。
比如说,那个叫 Frogs 的元胞自动机,规则按 Golly 的写法是 12/34/3。最后这个数字 3 表示它有三种状态:一个是死,一个是活,一个是正在死亡。也就是说,一个活细胞在临死前只能多活一个回合。前面的 12 说的是:一个活细胞要想继续好好活着,八个邻居里活细胞的个数必须是1或者2; 34 的意思则是:一个已经死掉的细胞要想活过来,周围的活细胞个数必须是3或者4。

Wolfram|Alpha 的元胞自动机应该也是 Generations 一类,但肉眼不容易看出具体是什么规则。不过不要紧,我们还有 Mathematica。直接看不出,我们就把图片二值化了看,拆成一个个连通分支来看,数清了每个点周围的活细胞个数再看。
当年的代码我没有留着。下面的代码都是新写的,用了不少这几年才引进的新函数。
首先当然是导入图片并去掉重复帧。我一开始没发现截图中有重复帧,导致结果很奇怪,还以为它不是 Generations 的规则。
CA = First /@ Split@Import["WolframAlphaCA.gif"]; |
先试着把第一帧二值化一下,发现它还有一个框框:
Binarize[CA[[1]]] |

摘掉黑框再二值化。二值化的阈值不能太低,以免最小的点消失;也不能太高,以免相邻的点连起来。试了好几个数,0.85 是最合适的。
Binarize[ImagePad[#, -BorderDimensions@#], 0.85] &@CA[[1]] |

看起来不错。于是我就把每一帧都二值化了。顺手还给它们反个色,以方便下一步处理。
CABinarized = 1 - Binarize[ImagePad[#, -BorderDimensions@#], 0.85] & /@ CA; |
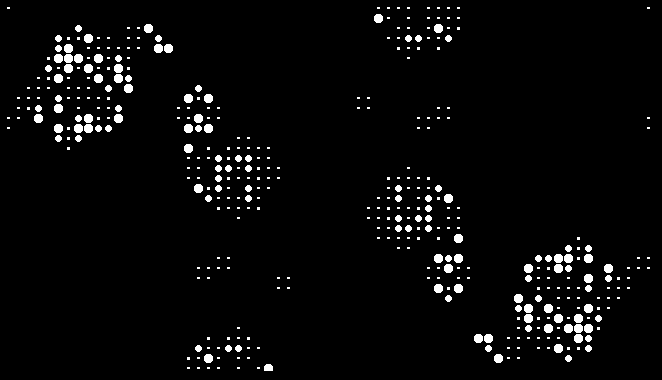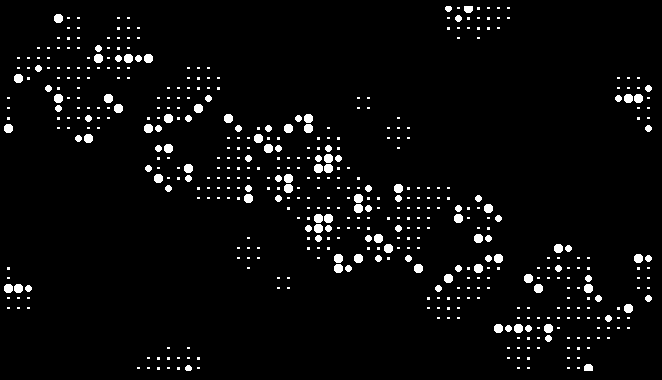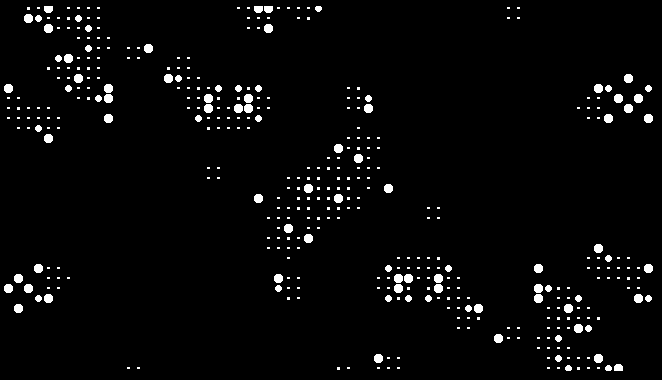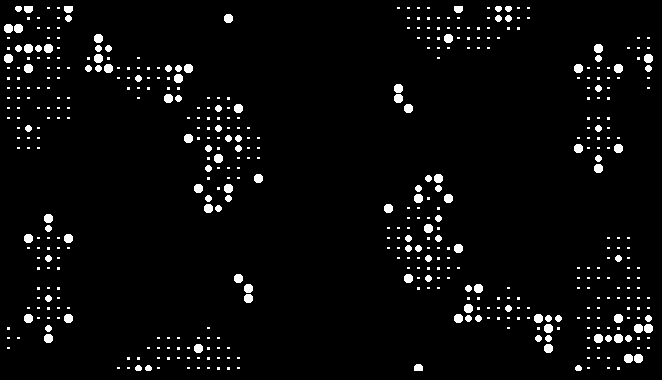
现在每个细胞——死细胞不算——是这个图片的一个连通分支。细胞的状态对应于连通分支的大小。于是可以用上 ComponentMeasurements 函数。
先来看看这些细胞的位置。还是先看第一帧,算出所有连通分支的重心,把所有的横坐标和所有的纵坐标分别从小到大排列:
Union /@ Transpose[Last /@ ComponentMeasurements[CABinarized[[1]], "Centroid"]] |
结果是:
{{8.5, 18.5, 28.5, 38.5, 48.5, 58.5, 68.5, 78.5, 88.5, 98.5, 108.5, |
可以看出相邻的两个点之间大概差10个像素,整个图片中的细胞个数是 65 × 37。这里出现了一个很奇怪的 12.2193,是因为最下面一排有一个点只露出了大半。为了避免这种问题,我去掉了最外面的一圈细胞,只取重心的横坐标在 15 和 645 之间,纵坐标在 15 和365 之间的那些连通分支。
然后看连通分支的大小。取了一下 Union,发现图片的每一帧,在去掉了最外面的一圈细胞之后,连通分支的大小都只有 {6, 9, 37, 69} 这么四种:
Union[Last /@ |
于是这四种不同大小的活细胞和濒死的细胞,再加上死细胞,一共有五种状态。最初的截图里分不太清的两种状态,在这里分别对应 6 和 9。
然后我们可以把这些图片化成代表细胞状态的数组,把 {6, 9, 37, 69} 分别换成 {1, 2, 3, 4},死细胞还是用 0 表示。
CAArray = |
如果它真的是 Generations 的规则,每个细胞在下一回合的状态就完全由它在这一回合的状态和周围的活细胞的个数来决定。于是来算一算,看看是否如此:
Union@Flatten@ |
结果是:
{{0, 0} -> 0, {0, 1} -> 0, {0, 2} -> 0, {0, 3} -> 1, {0, 4} -> 0, |
结果中的这些数据是 {本回合的状态, 周围的活细胞个数} -> 下一回合的状态。比如说, {1, 3} -> 2 表示某个细胞的状态是 1,也就是一个活细胞;它周围的活细胞个数,包括它本身,是 3;它在下一回合的状态是 2,也就是说刚刚开始死亡。这里同样的键总是对应同样的值,因此每个细胞在下一回合的状态确实由这两个数来决定。
我们还能看出,0 只会变成 0 或 1, 1 只会变成 1 或 2,2 只会变成 3,3 只会变成 4,4 只会死亡。
果然是 Generations。
再来算算这个规则在 Golly 里该怎么写:
{Cases[%, ({1, x_} -> 1) :> x - 1], Cases[%, ({0, x_} -> 1) :> x], |
结果是:
{{3, 4, 5, 7}, {3, 5, 7}, 5} |
也就是说它在 Golly 里叫做 3457/357/5。
这个规则的含义是:它有五种状态。0 表示死,1 表示活,2、3、4 都是死亡的过程。每个回合每个细胞的变化如下:
0-> 如果相邻的活细胞个数是3、5、7,则变成1,否则还是01-> 如果相邻的活细胞个数是3、4、5、7,则保持是1,否则变成22->33->44->0
知道了规则,就可以把它放到 Golly 里,乱搞一通。一旦找到了好玩的东西,还可以用 Mathematica 来制成 GIF。这就是下一篇文章的内容了。